Bassa variabilità del DNA satellitare nelle popolazioni naturali di Drosophila antonietae coinvolte in diversi eventi evolutivi
Traduzione automatica
L'articolo originale è scritto in lingua EN (link per leggerlo) .
Drosophila antonietae è una specie cactofila che si trova nella foresta mesofila del bacino del fiume Paraná–Paraguay e nelle dune della costa sud-atlantica del Brasile. Sebbene la struttura genetica delle popolazioni del bacino del fiume Paraná–Paraguay sia già stata stabilita, la relazione tra queste popolazioni e quelle sulla costa atlantica è controversa. In questo studio, abbiamo confrontato 33 unità ripetitive di DNA satellitare pBuM-2 isolate da individui di 8 popolazioni di D. antonietae in queste regioni geografiche, comprese alcune popolazioni trovate all'interno di una zona di contatto con il vicino D. serido. Le sequenze pBuM-2 hanno mostrato una bassa variabilità interpopolazionale. Questo risultato è stato interpretato come una conseguenza sia del flusso genico tra le popolazioni sia del crossing over disuguale che promuove l'omogeneizzazione degli array in tandem. I risultati presentati qui, insieme a quelli di studi precedenti, evidenziano l'uso di pBuM-2 per risolvere conflitti tassonomici all'interno del gruppo di specie D. buzzatii.
Introduzione
Drosophila antonietae è un membro del gruppo di specie D. buzzatii, un gruppo monofiletico composto da 7 specie sorelle naturalmente endemiche della regione neotropicale che si trovano nelle foreste aperte e secche dell'est dell'America del Sud (Manfrin e Sene 2006). Queste specie utilizzano tessuti necrotici di cactus come siti di riproduzione (Pereira et al. 1983); per questo motivo, il loro areale geografico è ristretto all'area di distribuzione dei cactus ospiti. Pertanto, D. antonietae è un modello interessante per studiare i processi evolutivi (Manfrin e Sene 2006).
La distribuzione geografica di D. antonietae può essere caratterizzata in 2 modi. Nelle valli racchiuse dal bacino idrografico del fiume Paraná–Paraguay, le popolazioni si trovano in foreste mesofile associate al cactus Cereus hildemannianus (Tidon-Sklorz e Sene 2001; Mateus e Sene 2003). Nella sua distribuzione sud-est, D. antonietae si trova nelle regioni di dune della costa atlantica, utilizzando principalmente C. hildemannianus ma anche cactus del genere Opuntia (Ruiz et al. 2000; Manfrin e Sene 2006) (Figura 1).
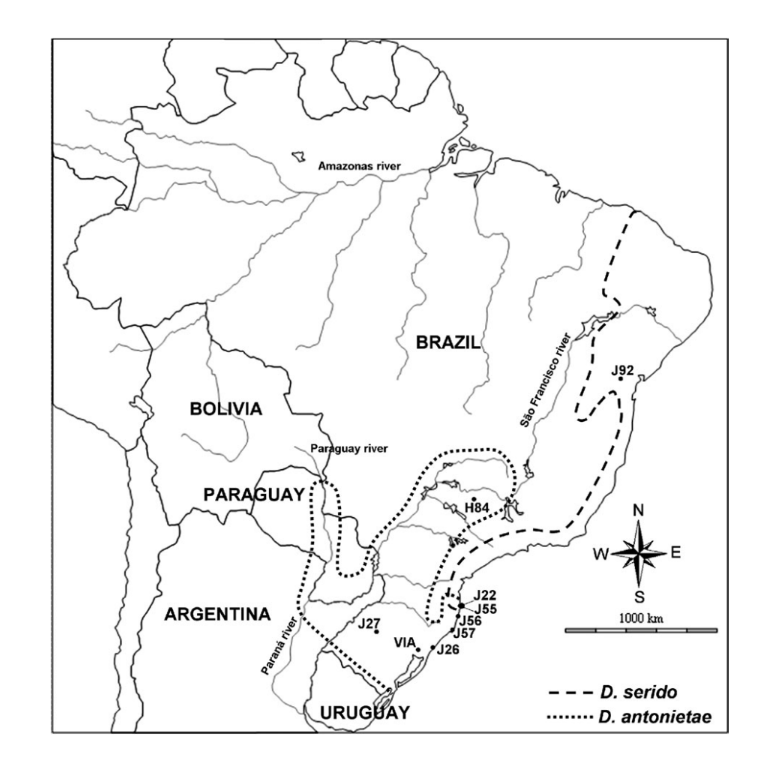
Studi precedenti hanno suggerito che le popolazioni di D. antonietae del bacino fluviale Paraná–Paraguay sono strutturate secondo il modello di isolamento per distanza a causa della correlazione positiva tra le distanze morfologiche e genetiche con le distanze geografiche ed ecologiche (quest'ultime definite come la distanza tra 2 popolazioni lungo il corso del fiume) (de Brito et al. 2002; Mateus e Sene 2007). Basandosi sul numero di migranti per generazione, Mateus e Sene (2007) hanno suggerito un effetto moderato sia del flusso genico che della deriva genetica in queste popolazioni di D. antonietae, con il flusso genico che si sovrappone alla deriva genetica promuovendo un gradiente genetico.
Un'analisi filogeografica basata sul DNA mitocondriale ha suggerito che la distribuzione sud-est di D. antonietae, sulla costa atlantica del Brasile, è stata colonizzata da eventi di espansione delle popolazioni di D. antonietae dalla depressione centrale dello stato di Rio Grande do Sul (de Brito et al. 2002; Manfrin e Sene 2006). Tuttavia, questa analisi non è riuscita a determinare se questa espansione fosse seguita da un mantenimento del flusso genico, con acquisizione di struttura della popolazione secondo il modello di isolamento per distanza, o seguita da frammentazione, implicando un'assenza di flusso genico tra le popolazioni della costa atlantica e quelle del bacino fluviale Paraná–Paraguay (de Brito et al. 2002). I dati sull'inversione cromosomica potrebbero suggerire un certo grado di frammentazione tra le popolazioni di questi 2 ambienti diversi, poiché quelle popolazioni delle valli del bacino fluviale Paraná–Paraguay sono polimorfiche per le inversioni 2y8, 2z8 e 5e, mentre le popolazioni della costa atlantica sono polimorfiche per 2z8 (Ruiz et al. 2000). Le popolazioni della costa atlantica di D. antonietae hanno un'altra caratteristica importante: sono in contatto con la specie strettamente correlata D. serido (D. buzzatii cluster) (Ruiz et al. 2000; Manfrin e Sene 2006). Quest'area di sovrapposizione è il risultato di D. serido che si espande dalla costa atlantica settentrionale a quella meridionale e condivide un'area geografica con
D. antonietae popolazioni (de Brito et al. 2002; Manfrin e Sene 2006) (Figura 1). I tratti morfologici e le analisi del DNA mitocondriale suggeriscono che l'ibridazione tra
D. antonietae e D. serido sia avvenuta all'interno della zona di contatto (C.K.B. Santos e colleghi, dati non pubblicati).
Il DNA satellitare è costituito da sequenze di DNA altamente ripetitive disposte in tandem che rappresentano una componente sostanziale dell'eterocromatina degli organismi eucarioti (Charlesworth et al. 1tt4; Palomeque e Lorite 2008). I satelliti si trovano tipicamente vicino alle regioni centromeriche e, meno frequentemente, nei telomeri (Ugarkovic e Plohl 2002; Kuhn et al. 2008). Queste sequenze evolvono in modo concertato, mentre i principali meccanismi molecolari coinvolti nella loro evoluzione sono la replicazione per slittamento, il crossing over disuguale, la conversione genica e la replicazione a cerchio rotolante (Dover 1982; Charlesworth et al. 1994).
Alcune delle famiglie di DNA satellitare evolvono rapidamente, consentendo il loro utilizzo come marcatori molecolari negli studi comparativi interspecifici (Watabe et al. 1997; Picariello et al. 2002; Kuhn e Sene 2005). Tuttavia, l'uso di queste sequenze come marcatori molecolari negli studi di popolazione è stato relativamente poco esplorato, nonostante il fatto che l'evoluzione concertata sia stata riportata in popolazioni di Cyprinodon variegates (Cyprinodontidae) (Elder e Turner 1994) e Acrossocheilus paradoxus (Cyprinidae) (Wu et al. 1999).
Tre famiglie di DNA satellitare condivise tra le specie del cluster D. buzzatii sono state formalmente descritte: pBuM (Kuhn et al. 1999, 2008; Kuhn e Sene 2005), DBC-150 (Kuhn et al. 2007), e SSS139 (Franco et al. 2008). La famiglia pBuM è composta da 2 sottofamiglie, pBuM-1 e pBuM-2 (Kuhn e Sene 2005; Kuhn et al. 2008). La sottofamiglia pBuM-2 è composta da monomeri ricchi di AT di 370 bp, che presentano differenze qualitative (sostituzione nucleotidica fissa) e quantitative (numero di copie) tra le specie del cluster D. buzzatii (Kuhn e Sene 2005; Kuhn et al. 2007, 2008).
In questo lavoro, dimostriamo la variabilità intraspecifica del DNA satellitare pBuM-2 in D. antonietae, utilizzando popolazioni provenienti da 2 ambienti diversi (il bacino Paraná–Paraguay e la costa atlantica) che presentano storie evolutive distinte, associate al mantenimento del flusso genico nel primo e a eventi diibridazione con la specie strettamente correlata D. serido nel secondo. Abbiamo scelto di utilizzare il DNA satellitare pBuM come marcatore molecolare perché presenta 14 sostituzioni nucleotidiche diagnostiche tra D. antonietae e D. serido (Kuhn e Sene 2005). Pertanto, pBuM consente l'identificazione tassonomica di esemplari isolati dalla zona di contatto. Inoltre, poiché le dinamiche evolutive delle sequenze di DNA satellitare a livello intraspecifico non sono completamente comprese, ulteriori dati empirici potrebbero contribuire a una migliore comprensione del processo sottostante l'evoluzione del DNA satellitare (Ugarkovic e Plohl 2002; Palomeque e Lorite 2008).
Materiali e Metodi
Campioni
Abbiamo studiato 8 popolazioni brasiliane di D. antonietae (Figura 1). Tre di queste popolazioni si trovano nei vallate del bacino del fiume Paraná–Paraguay: J2/ (Santiago, stato di Rio Grande do Sul), Via (Viamão, stato di Rio Grande do Sul) e H84 (Serrana, stato di São Paulo, numero di accesso AY656609– AY656615; Kuhn e Sene 2005); e 5 si trovano nelle dune della costa atlantica, all'interno della zona di contatto con
D. serido: J56 (Garopaba, stato di Santa Catarina), J55 (spiaggia di Armaxcão, Florianópolis, stato di Santa Catarina), J22 (spiaggia di Joaquina, Florianópolis, stato di Santa Catarina), J5/ (Laguna, stato di Santa Catarina) e J26 (Osório, stato di Rio Grande do Sul) (Figura 1). Le sequenze di D. antonietae sono state analizzate insieme alle sequenze G pBuM-2 di D. serido (popolazione Jf2—Milagres, stato di Bahia, numero di accesso AY656616–AY656621; Kuhn e Sene 2005), al fine di identificare alcuni eventi di introgressione nelle popolazioni della costa atlantica.
Isolamento del DNA satellitare pBuM-2 e analisi della sequenza
Il DNA genomico da un individuo maschile (mosche catturate in natura) per popolazione è stato estratto utilizzando il Wizard Genomic DNA Purification Kit (Promega). Il DNA genomico è stato sottoposto a reazione a catena della polimerasi per amplificare le copie della famiglia pBuM-2 [primer A2F (CGGAGTA-TTTTTCATTCGAC) e A2R (GGTATGCCATAAAG-AAGTCG)] secondo Kuhn et al. (2008). Le bande risultanti di circa 400 bp sono state eluite dal gel mediante incubazione notturna in una soluzione di eluzione (500 mM NaAc; 1 mM acido etilendiamminotetraacetico). I frammenti recuperati sono stati clonati utilizzando il kit di clonazione a estremità smussate pMOSBlue (RPN 5110; Amersham Pharmacia Biotech). Il DNA plasmidico è stato preparato seguendo la metodologia descritta in Sambrook et al. (1989), e la reazione del template DNA per il sequenziamento è stata preparata secondo il manuale del BigDye Terminator Cycle Sequencing Ready Reaction Kit (PerkinElmer). Il sequenziamento automatico del DNA è stato eseguito su un sequenziatore ABI Prism 377 (PerkinElmer).
Gli allineamenti delle sequenze sono stati effettuati in CLUSTALW 1.8 (Thompson et al. 1994) e modificati in BioEdit (Hall 1999). Abbiamo utilizzato l'algoritmo e il modello statistico proposto da Betrán et al. (1997) per rilevare i tratti di conversione genica. Il parametro Rm, che stima il numero minimo di eventi di ricombinazione in un campione di DNA (Hudson e Kaplan 1985), è stato utilizzato per rilevare eventi di ricombinazione. Sebbene questo parametro non misuri specificamente il crossing over disuguale, è ragionevole assumere che il Rm avrebbe valori superiori a zero se si fosse verificato un crossing over disuguale. In questo senso, utilizziamo Rm come prova indiretta di crossing over disuguale. Le distanze genetiche sono state calcolate secondo il p-distanza, che è la proporzione delle differenze nucleotidiche tra 2 sequenze (cioè, p 5 Np/N, dove Np è la differenza dei nucleotidi e N è il numero totale di nucleotidi). Sono stati applicati anche modelli di distanza più complessi ai dati, con risultati simili a quelli presentati dalla p-distanza (dati non mostrati). La matrice delle distanze è stata utilizzata per creare un fenogramma utilizzando il metodo del neighbor joining (Saitou e Nei 1987). I calcoli delle distanze genetiche e i fenogrammi sono stati effettuati utilizzando il programma MEGA 3.0 (Kumar et al. 2004).
Abbiamo anche investigato i modelli di variazione intraspecifica in D. antonietae mediante analisi della varianza molecolare (AMOVA; Excoffier et al. 1992). Per l'AMOVA, le popolazioni di D. antonietae sono state raggruppate in 2 gruppi: costa atlantica e bacino del fiume Paraná–Paraguay. L'AMOVA è stata eseguita utilizzando Arlequin 3.0 (Excoffier et al. 2005).
Risultati e Discussione
Sono stati ottenuti ventisei nuovi monomeri di DNA satellitare pBuM-2: 4 dalla località J56 (numero di accesso FJ935973– FJ93597G); 4 dalla località Via (numero di accesso FJt35t77–FJt35t80); 4 dalla località J26 (numero di accesso FJ935981–FJ935984); 3 dalla località J5/ (numero di accesso FJ935985–FJ935987); 5 dalla località J2/ (numero di accesso FJ935988–FJ935992); 2 dalla località J55 (numero di accesso FJ935993–FJ935994); e 4 dalla località J22 (numero di accesso FJ935995–FJ935998). L'alta somiglianza nucleotidica tra le sequenze ottenute in questo lavoro e quelle precedentemente descritte (Kuhn e Sene 2005) conferma che appartengono alla famiglia di DNA satellitare pBuM-2.
Tutte le sequenze ottenute presentano la struttura primaria tipica di D. antonietae e sono state raggruppate nello stesso ramo nel fenogramma, insieme alle sequenze pBuM-2 precedentemente descritte di D. antonietae (Figura 2), anche quelle delle popolazioni all'interno della zona di contatto con D. serido, dove è stata suggerita l'ibridazione tra D. antonietae e D. serido con introgressione di DNA mitocondriale (C.K.B Santos, F.M. Sene e M.H. Manfrin, dati non pubblicati). Questa situazione potrebbe essere correlata alle osservazioni che i geni mitocondriali tendono a introgressare più facilmente rispetto alle sequenze nucleari (Dorado et al. 1992; Arnold 1993). Le spiegazioni più citate per questa osservazione sono legate ai processi di disequilibrio cito-nucleare (Arnold 1993) e alla sterilità dei maschi ibridi (Aubert e Solignac 1990).
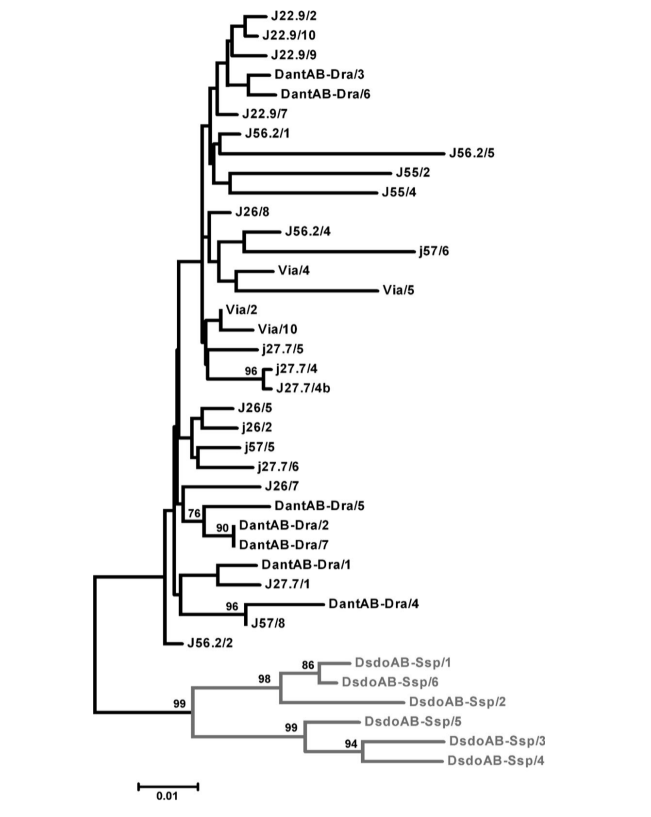
C'è un alto livello di somiglianza di sequenza tra le sequenze pBuM-2 di D. antonietae (95,7% in media), e non sono state trovate mutazioni diagnostiche per una popolazione particolare. La variabilità interpopolazionale era molto simile alla variabilità intrapopolazionale (Tabella 1), e le sequenze della stessa popolazione non si sono raggruppate insieme in un ramo specifico nel dendrogramma (Figura 2). L'AMOVA eseguita con le popolazioni suddivise per gruppi geografici (bacino del fiume Paraná–Paraguay e costa atlantica) ha mostrato che il 99,30% della diversità genetica totale poteva essere attribuita alla variabilità intragruppo, mentre solo lo 0,70% era attribuito alla variabilità intergruppo (Tabella 2). Il Фst calcolato era basso e non aveva supporto statistico (Tabella 2).
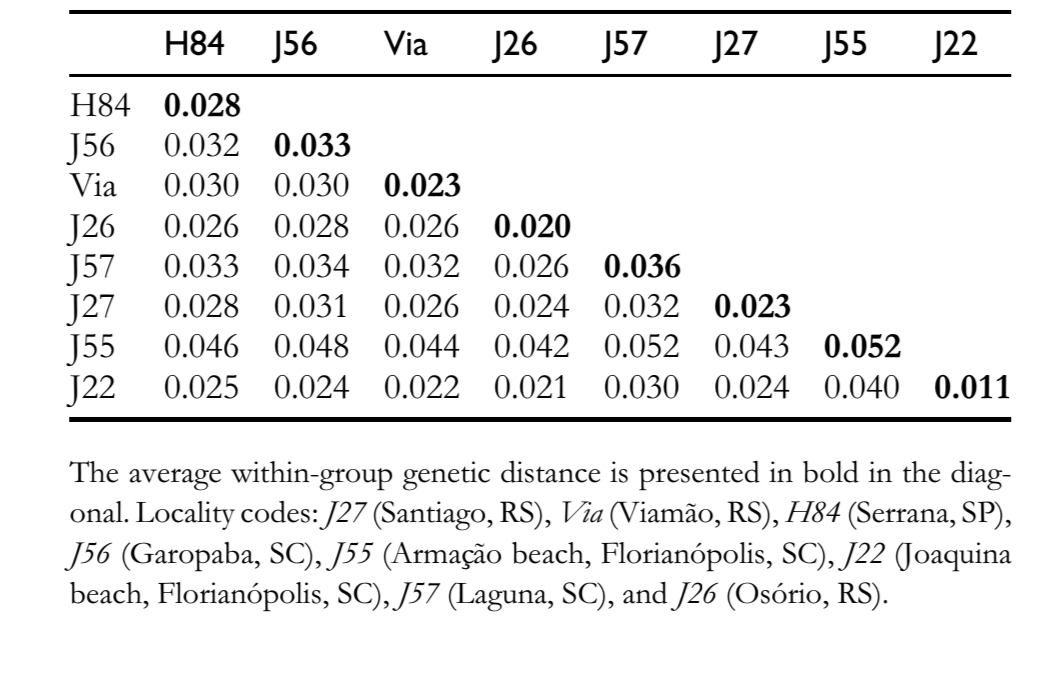

Le popolazioni di D. antonietae del bacino del fiume Paraná–Paraguay sono strutturate secondo un modello di isolamento per distanza (Monteiro e Sene 1995; Mateus e Sene 2007). Le inversioni cromosomiche osservate suggeriscono una frammentazione tra le popolazioni della costa atlantica e quelle del bacino del fiume Paraná–Paraguay (Ruiz et al. 2000), mentre i dati del DNA mitocondriale non sono riusciti a concludere se la frammentazione o l'isolamento per distanza fosse la struttura dominante che definisce la relazione di questi 2 gruppi di popolazione (de Brito et al. 2002). L'alta somiglianza nucleotidica di pBuM-2 in D. antonietae (Figura 2 e Tabelle 1 e 2) è congruente con il mantenimento del flusso genico tra le popolazioni di questa specie, anche tra quelle allocate nel bacino del fiume Paraná–Paraguay e la costa atlantica. I nostri risultati suggeriscono che l'isolamento per distanza dovrebbe spiegare la variabilità del DNA mitocondriale in D. antonietae; questo modello prevede flusso genico tra le popolazioni, come inferito sulla base dei dati del DNA satellite presenti qui. In questo contesto, i più bassi polimorfismi di inversione cromosomica nelle popolazioni della costa atlantica di D. antonietae, rilevati da Ruiz et al. (2000), potrebbero essere spiegati come una conseguenza di eventi storici e demografici come l'espansione della popolazione dalle popolazioni delle valli del bacino del fiume Paraná–Paraguay, come suggerito dal DNA mitocondriale (de Brito et al. 2002). In alternativa, questa differenziazione della popolazione potrebbe essere associata all'adattamento locale a ambienti distinti in presenza di flusso genico, poiché le inversioni cromosomiche possono essere correlate a caratteristiche adattative in Drosophila cactofila (Hasson et al. 1992; Fernández Iriarte et al. 2003). Un'altra spiegazione per l'alta conservazione nucleotidica di pBuM-2 in D. antonietae si basa sulle dinamiche evolutive delle sequenze di DNA satellite. I meccanismi molecolari legati all'omogeneizzazione delle sequenze disposte in tandem, come il crossing over disuguale e la conversione genica attraverso un processo chiamato drive molecolare (Dover 1982; Strachan et al. 1985), potrebbero agire sul DNA satellite pBuM in questa specie. Le nostre analisi non hanno identificato tratti di conversione genica nelle sequenze pBuM-2 isolate da D. antonietae, suggerendo che questo meccanismo molecolare non influisce sulla diversità delle sequenze pBuM-2 in questa specie. Tuttavia, è stato rilevato un minimo di 4 eventi di ricombinazione nel campione (Rm = 4) tra gli intervalli di 130–136, 136–155, 174–376 e 376–378. Questi eventi di ricombinazione potrebbero essere una prova indiretta di crossing over disuguale che contribuisce al mantenimento della struttura primaria delle sequenze pBuM-2 nelle diverse popolazioni di D. antonietae. Coerente con questa ipotesi, il genoma di D. antonietae contiene un gran numero di monomeri pBuM-2 (Kuhn et al. 2007; Kuhn et al. 2008), un fatto che aumenta la probabilità di crossing over disuguale (Smith1976), e di conseguenza omogeneizzazione ed evoluzione concertata.
Queste 2 ipotesi alternative che spiegano l'alta somiglianza nucleotidica trovata nei monomeri di pBuM-2 di D. antonietae non sono mutualmente esclusive. In questo contesto, proponiamo che un equilibrio tra il flusso genico tra le popolazioni e gli eventi di omogeneizzazione degli array di DNA satellitare, promossi da crossing over disuguale, contribuisca alla conservazione della struttura primaria di pBuM-2 nelle specie di D. antonietae .
L'assenza di divergenza molecolare nelle sequenze di DNA satellitare tra popolazioni della stessa specie, come trovato nel presente articolo, è stata osservata anche in altre specie di insetti (Bachmann et al. 1994, 1998; Lorite et al. 2002; Feliciello et al. 2005). Ad esempio, Bachmann et al. (1994) hanno trovato alti livelli di somiglianza genetica tra i monomeri del pDoP102 satellitare in popolazioni di Dolichopoda schiavazzii (Ortotteri), i cui dati allozimatici indicano che sono stati isolati l'uno dall'altro per almeno 235 000 anni. Secondo questi autori, l'omogeneità in pDoP102 anche in popolazioni isolate potrebbe essere dovuta alla biologia del DNA satellitare perché il crossing over disuguale e la conversione genica possono indurre il mantenimento della struttura primaria delle sequenze di DNA satellitare (Bachmann et al. 1994). In un altro studio, la presenza di tratti di conversione genica nei monomeri di una famiglia di DNA satellitare è stata utilizzata come prova di eventi di omogeneizzazione in questo DNA satellitare, spiegando l'alta conservazione intraspecifica del DNA satellitare trovata nel coleottero fitofago Xanthogaleruca luteola (Lorite et al. 2002).
Nel D. buzzatii cluster di specie, è stata identificata anche un'alta conservazione intraspecifica della sequenza pBuM-2 in D. seriema (Kuhn e Sene 2004) e D. gouveai (Franco, Kuhn, et al. 2006). Inoltre, le popolazioni di D. buzzatii sono omogenee per quanto riguarda il DNA satellitare pBuM-1, un altro membro della famiglia pBuM (Kuhn et al. 2003). In D. seriema e D. buzzatii, si è suggerito che la mancanza di differenziazione delle popolazioni nelle sequenze di DNA satellitare derivi da eventi di flusso genico passati e attuali tra le popolazioni, che sono compatibili con i risultati ottenuti da altri marcatori genetici analizzati nelle stesse popolazioni (Kuhn et al. 2003; Kuhn e Sene 2004). Per
D. gouveai, che è strutturato secondo un modello di ‘‘isola’’ (de Brito et al. 2002; Moraes e Sene 2007), l'ipotesi principale per spiegare la conservazione della struttura primaria nel DNA satellitare pBuM-2 era che meccanismi di omogeneizzazione indipendenti a favore delle unità ripetitive condivise tra le popolazioni si fossero verificati dopo l'evento di isolamento delle popolazioni di D. gouveai (Franco, Kuhn, et al. 2006). Il cluster di D. buzzatii è composto da specie sorelle con tassonomia basata sulle caratteristiche morfometriche dell'aedeago, che è l'organo intromittente dei genitali maschili degli insetti (Vilela 1983; Tidon-Sklorz e Sene 2001; Franco, Prado, et al. 2006). Pertanto, l'identificazione degli individui femminili del cluster D. buzzatii, forniti dall'ambiente naturale, avviene attraverso l'identificazione della loro prole maschile rendendo necessaria l'istituzione di linee isofemmina, che può essere un compito arduo per le specie di Drosophila cactofile. Inoltre, la corretta discriminazione delle specie basata solo su caratteri quantitativi, come la morfologia dell'aedeago, a volte è difficile a causa della presenza di individui morfologicamente ambigui, specialmente quando gli individui vengono raccolti all'interno delle zone di contatto tra le specie del cluster D. buzzatii. In questo senso, la forte conservazione intraspecifica delle sequenze di DNA satellitare pBuM ottenute in diversi studi (Kuhn et al. 2003; Kuhn e Sene 2004; Franco, Kuhn, et al. 2006; presente articolo), anche in specie geneticamente strutturate, evidenzia l'utilità di pBuM-2 nella risoluzione dei conflitti tassonomici all'interno delle specie del cluster D. buzzatii, un modello riconosciuto per studi di biologia evolutiva (Manfrin e Sene 2006).
Autori: Fernando Faria Franco, Fabio Melo Sene, Maura Helena Manfrin
Riferimenti:
- Arnold J. 1993. Disequilibri cytonucleari nelle zone ibride. Annu Rev Ecol Syst. 24:521–554.
- Aubert J, Solignac M. 1990. Evidenza sperimentale per l'introgressione del DNA mitocondriale tra specie di Drosophila. Evoluzione. 44:1272–1282.
- Bachmann L, Tomiuk J, Adis J, Vohland K. 1998. Differenziazione genetica del millepiedi Pycnotropis epiclysmus che abita foreste amazzoniche stagionalmente allagate e non allagate. J Zool Syst Evol Res. 36:65–70.
- Bachmann L, Venanzetti F, Sbordoni V. 1994. Caratterizzazione di una famiglia di DNA satellitare specifica per specie di Dolichopoda schiavazzi (Orthoptera, Rhaphidophoridae) grilli delle grotte. J Mol Evol. 39:274–281.
- Betrán E, Rozas J, Navarro A, Barbadilla A. 1997. La stima del numero e della distribuzione della lunghezza dei tratti di conversione genica da dati di sequenza DNA di popolazione. Genetics. 146:89–99.
- Charlesworth B, Sniegowski P, Stephan W. 1tt4. Le dinamiche evolutive del DNA ripetitivo negli eucarioti. Nature. 371:215–220.
- de Brito AR, Manfrin MH, Sene FM. 2002. Analisi cladistica nidificata delle popolazioni brasiliane di Drosophila serido. Mol Phylogenet Evol. 22: 131–143.
- Dorado O, Rieseberg LH, Arias D. 1992. Introgressione del DNA cloroplastico nei girasoli della California meridionale. Evoluzione. 46:566–572.
- Dover G. 1982. Drive molecolare: una modalità coesa di evoluzione delle specie. Nature. 299:111–117.
- Elder JF, Turner BJ. 1tt4. Evoluzione concertata a livello di popolazione: sequenze di DNA satellitare HindIII dei pesci pupfish. Proc Natl Acad Sci U S A. 91: 994–998.
- Excoffier L, Laval G, Schneider S. 2005. Arlequin (versione 3.0): un pacchetto software integrato per l'analisi dei dati di genetica delle popolazioni. Evol Bioinform Online. 1:47–50.
- Excoffier L, Smouse PE, Quattro JM. 1992. Analisi della varianza molecolare inferita da distanze metriche tra haplotipi DNA: applicazione ai dati di restrizione del DNA mitocondriale umano. Genetics. 131:479–491.
- Feliciello I, Picariello O, Chinali G. 2005. La prima caratterizzazione della variabilità complessiva delle unità ripetitive in una specie rivela caratteristiche inaspettate del DNA satellitare. Gene. 349:153–164.
- Fernández Iriarte PJ, Norry FM, Hasson ER. 2003. Gli inversioni cromosomiche influenzano la dimensione e la forma del corpo in diverse risorse riproduttive in Drosophila buzzatii. Eredità. 91:51–59.
- Franco FF, Kuhn GCS, Sene FM, Manfrin MH. 2006. Conservazione delle sequenze di DNA satellitare pBuM–2 tra popolazioni geograficamente isolate di Drosophila gouveai dal Brasile. Genetica. 128:287–295.
- Franco FF, Prado PRR, Sene FM, Costa LF, Manfrin MH. 200G. Morfologia dell'aedeago come marcatore discriminante in due specie cactofile strettamente correlate di Drosophila (Diptera; Drosophilidae) in Sud America. An Acad Bras Cienc. 78:203–212.
- Franco FF, Sene FM, Manfrin MH. 2008. Caratterizzazione molecolare della famiglia di DNA satellitare SSS13t in specie sorelle del cluster Drosophila buzzatii. Genet Mol Biol. 31:155–159.
- Hall TA. 1999. BioEdit: un editor di allineamento di sequenze biologiche user-friendly e programma di analisi per Windows t5/t8/NT. Nucleic Acids Symp Ser. 41:95–98.
- Hasson E, Fanara JJ, Rodríguez C, Vilardi JC, Reig OA, Fontdevila A. 1992. La storia evolutiva di Drosophila buzzatii. XXIV. Le inversioni del secondo cromosoma hanno effetti medi diversi sulla lunghezza del torace. Eredità. 68:557–563.
- Hudson RR, Kaplan NL. 1985. Proprietà statistiche del numero di eventi di ricombinazione nella storia di un campione di sequenze DNA. Genetics. 111:147–164.
- Kuhn GCS, Bollgönn S, Sperlich D, Bachmann L. 1999. Caratterizzazione di un DNA satellitare specifico per specie di Drosophila buzzatii. J Zool Syst Evol Res. 37:109–112.
- Kuhn GCS, Franco FF, Manfrin MH, Moreira-Filho O, Sene FM. 2007. Bassi tassi di omogeneizzazione della famiglia di DNA satellitare DBC-150 ristretta a una sola coppia di microcromosomi in specie del cluster Drosophila buzzatii. Chromosome Res. 15:457–469.
- Kuhn GCS, Franco FF, Silva WA Jr, Martinez-Rossi NM, Sene FM. 2003. Sulla variabilità del DNA satellitare pBuM18t tra popolazioni sudamericane di Drosophila buzzatii. Hereditas. 139:161–166.
- Kuhn GCS, Sene FM. 2004. Caratterizzazione e variabilità interpopolazione di un complesso DNA satellitare HpaI di Drosophila seriema (gruppo repleta). Genetica. 121:241–249.
- Kuhn GCS, Sene FM. 2005. Turnover evolutivo di due sottogruppi di DNA satellitare pBuM nel cluster di specie Drosophila buzzatii (gruppo repleta): da alpha a alpha/beta array. Gene. 349:77–85.
- Kuhn GCS, Sene FM, Moreira-Filho O, Schwarzacher T, Heslop-Harrison JS. 2008. L'analisi delle sequenze, la distribuzione cromosomica e l'organizzazione a lungo raggio mostrano che il rapido turnover di nuovi e vecchi ripetizioni di DNA satellitare pBuM porta a diversi schemi di variazione in sette specie del cluster Drosophila buzzatii. Chromosome Res. 16:307–324.
- Kumar S, Tamura K, Nei M. 2004. MEGA3: software integrato per l'analisi genetica evolutiva molecolare e l'allineamento delle sequenze. Brief Bioinform. 5:150–163.
- Lorite P, Carrillo JA, Garneria I, Petitpierre E, Palomeque T. 2002. DNA satellitare nel coleottero delle foglie di olmo, Xanthogaleruca luteola (Coleoptera, Chrysomelidae): caratterizzazione, analisi interpopolazione e localizzazione cromosomica. Cytogenet Genome Res. 98:302–307.
- Manfrin MH, Sene FM. 200G. Drosophila cactofile in Sud America: un modello per studi evolutivi. Genetica. 126:57–75.
- Mateus RP, Sene FM. 2003. Variazione temporale e spaziale degli allozimi nella Drosophila cactofile sudamericana Drosophila antonietae (Diptera; Drosophilidae). Biochem Genet. 41:219–233.
- Mateus RP, Sene FM. 2007. Studio genetico di popolazione della variazione degli allozimi in popolazioni naturali di Drosophila antonietae (Insecta, Diptera). J Zool Syst Evol Res. 45:136–143.
- Monteiro SG, Sene FM. 1tt5. Studio morfometrico delle popolazioni di Drosophila serido delle regioni Centrale e Sud del Brasile. Rev Bras Genet (Suppl). 18:283.
- Moraes EM, Sene FM. 2007. Variazione microsatellitare e morfometrica in Drosophila gouveai: l'importanza relativa di fattori storici e attuali nella formazione della struttura genetica della popolazione. J Zoolog Syst Evol Res. 45:336–344.
- Palomeque T, Lorite P. 2008. DNA satellitare negli insetti: una revisione. Eredità. 100:564–573.
- Pereira MAQR, Vilela CR, Sene FM. 1983. Note sui siti di riproduzione e alimentazione di alcune specie del gruppo repleta del genere Drosophila (Diptera, Drosophilidae). Cienc Cult. 35:1313–1319.
- Picariello O, Feliciello I, Bellinero R, Chinali G. 2002. DNA satellitare S1 come marcatore tassonomico nelle rane brune: evidenza molecolare che Rana graeca graeca e Rana graeca italica sono specie diverse. Genome. 45: 63–70.
- Ruiz A, Cassian AM, Kuhn GCS, Alves MAR, Sene FM. 2000. Il puzzle della speciazione di Drosophila serido: mettere insieme nuovi pezzi. Genetica. 108:217–227.
- Saitou N, Nei M. 1987. Il metodo del neighbor-joining: un nuovo metodo per ricostruire alberi filogenetici. Mol Biol Evol. 4:406–425.
- Sambrook J, Fritsh EF, Maniatis T. 1989. Clonazione molecolare: un manuale di laboratorio. 2a ed. Cold Spring Harbor (NY): Cold Spring Harbor Laboratory Press.
- Smith GP. 1976. Evoluzione delle sequenze di DNA ripetute mediante crossover disuguale. Science. 191:528–535.
- Strachan T, Webb D, Dover GA. 1985. Fasi di transizione del drive molecolare in famiglie di DNA a copia multipla in Drosophila. EMBO J. 4:1701–1708.
- Thompson JD, Higgins DG, Gibson TJ. 1994. CLUSTAL W: migliorare la sensibilità dell'allineamento progressivo di sequenze multiple attraverso la pesatura delle sequenze, penalità specifiche per posizioni e scelta della matrice di pesi. Nucleic Acids Res. 22:4673–4680.
- Tidon-Sklorz R, Sene FM. 2001. Due nuove specie del set di sorelle di Drosophila serido (Diptera, Drosophilidae). Iheringia. 90:141–146.
- Ugarkovic D, Plohl M. 2002. Variazione nei profili di DNA satellitare—cause ed effetti. EMBO J. 21:5955–5959.
- Vilela CR. 1983. Una revisione del gruppo di specie Drosophila repleta (Diptera; Drosophilidae). Rev Bras Entomol. 27:1–114.
- Watabe H, Bachmann L, Haring E, Sperlich D. 1997. Studi tassonomici e molecolari su Drosophila sinobscura e D. hubeiensis, due specie sorelle del gruppo D. obscura. J Zool Syst Evol Res. 35:81–94.
- Wu WL, Wang JP, Tseng MC, Chiang TY. 1999. Clonazione e variabilità genetica di un DNA ripetitivo HindIII in Acrossocheilus paradoxos (Cyprinidae). Genome. 42:780–788.
