Variazione Genetica Nei Drosophila Hawaiani. IV. Somiglianza Alloenzimatica Tra D. Silvestris E D. Heteroneura Dell'Isola Di Hawaii
Traduzione automatica
L'articolo originale è scritto in lingua EN (link per leggerlo) .
Abstract
Le specie sono endemiche dell'isola più recente dell'arcipelago e sono ampiamente simpatiche. Sono facilmente distinguibili morfologicamente in entrambi i sessi. Utilizzando procedure elettroforetiche standard, abbiamo esaminato 25 loci che codificano per proteine strutturali da 539 silvestris e 325 heteroneura raccolti in tre località ampiamente separate dove le due specie sono simpatiche. I confronti a coppie all'interno e tra le specie mostrano i seguenti coefficienti di identità genetica (Nei's I): all'interno di silvestris 0.961 ± 0.01; all'interno di heteroneura 0.949 ± 0.02; tra silvestris e heteroneura, 0.939 ± 0.01. Né le differenze all'interno né tra le specie sono significative. Non ci sono differenze alleliche fisse né all'interno né tra le specie. Nelle tre aree di simpatria, le specie mostrano differenze di frequenza genica P < 0.05) in 9, 11 e 13 loci rispettivamente. Questo non è molto diverso dalla variazione all'interno di una delle specie nelle tre località. Le due specie hanno livelli simili di eterozigosi (H) (silvestris, 0.083; heteroneura, 0.089) e percentuale di loci polimorfici (entrambi 0.37). Si suggerisce che nonostante la loro divergenza morfologica, queste specie siano molto più recentemente formate rispetto alle classiche specie sorelle. Differenze allozimiche significative potrebbero non aver avuto il tempo di accumularsi.
DROSOPHILA silvestris e D. heteroneura sono una coppia di specie giganti strettamente correlate, endemiche solo alle foreste pluviali dell'isola di Hawaii, la più recente dell'arcipelago. Sono simpatiche nella maggior parte delle aree, tranne che ad altitudini più elevate dove le popolazioni tendono ad essere esclusivamente silvestris.
Le specie sono facilmente distinte morfologicamente (Hardy 1965) non solo per l'eccezionale testa larga in entrambi i sessi di heteroneura, ma anche per i modelli di colore del viso, del torace, delle pleure e dell'addome e per le differenze nel modello delle marcature delle ali. D. silvestris presenta sei inversioni uniche in stato polimorfico; una settima inversione è polimorfica in entrambe le specie (Carson e Stalker 1968; Craddock e Carson 1975). Non ci sono inversioni fisse tra le specie, e le due condividono un insieme comune di sequenze standard, cioè sono cromosomicamente omosequenziali.
Sebbene ci sia una forte isolamento sessuale tra le specie nei incroci di laboratorio (Ahearn et al. 1974; Kaneshiro 1976) F1, ibridi sono stati ottenuti da entrambi gli incroci reciproci (Craddok 1974; Ahearn e Val 1975). Entrambi i sessi di questi F1, ibridi sono completamente fertili e la segregazione genetica delle differenze morfologiche tra le specie è stata osservata nella generazione F e nei retroincroci (Val 1976).
Studi preliminari sugli allozimi hanno mostrato un'eccezionale somiglianza tra le due specie (S = 0.96; Johnson et al. 1975; Johnson e Carson 1975). Il presente articolo estende lo studio della variabilità genetica all'interno e tra le due specie a proteine solubili codificate da 25 loci. I dati provengono da tre aree di simpatria. I risultati confermano la straordinaria somiglianza biochimica delle specie nonostante le differenze genetiche tra di esse in morfologia, inversioni e comportamento.
Materiali e metodi
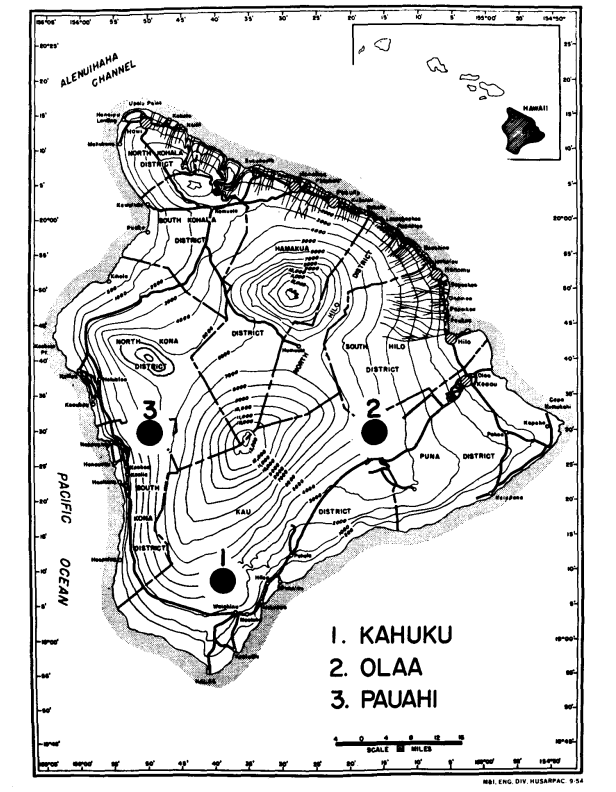
Nel 1974 e 1975, abbiamo raccolto le due specie da tre aree di elevazione moderata alle Hawaii (The Big Island) dove sono simpatriche. Queste sono (Figura 1): (1) Kahuku Ranch nella parte meridionale del distretto di Kau, (elevazione 1158 m); (2) Olaa, foresta vicino alla Volcano Experiment Station su Wright Road, distretto di Puna (elevazione 1238 m) e (3) Pauahi, distretto di South Kona (elevazione 1311 m). I maschi catturati di recente sono stati utilizzati per l'elettroforesi il prima possibile dopo la cattura. Le femmine sono state isolate e hanno prodotto prole F1 da utilizzare nelle preparazioni delle ghiandole salivari. Dopo aver prodotto larve, le madri sono state utilizzate per l'elettroforesi. Solo l'addome delle mosche è stato sottoposto a elettroforesi; il resto (testa, torace e appendici) è stato montato e collocato in una collezione da utilizzare per analisi morfologiche.
Dopo la rimozione, l'addome è stato congelato per una notte e poi omogeneizzato individualmente in 0,05 ml di acqua distillata deionizzata e ciascun supernatante assorbito da quattro stoppini di carta filtro Whatman No. I (1 X 0,2 cm). Questi stoppini sono stati quindi separati e applicati a quattro gel di amido orizzontali combinando diversi sistemi tampone. Dopo l'elettroforesi, ciascun gel è stato affettato orizzontalmente quattro o cinque volte, e queste fette sono state colorate separatamente per uno o più di 25 enzimi. Tutto il lavoro è stato svolto utilizzando Electrostarch lotto 303 a una concentrazione del 12%.
Le tre combinazioni di tampone utilizzate nello studio sono le seguenti: Sistema di tampone A = Tampone gel: pH 8.9, 0.0076 m Tris, 0.005 m citrato; Tampone elettrodi: pH 8.7, 0.269 m borato, 0.1 m idrossido di sodio. Sistema di tampone C = Tampone gel: pH 8.1, 0.074 m Tris, 0.008 m citrato; Tampone elettrodi: pH 8.1, catodo = 0.343 m Tris, 0.079 m citrato; anodo = 0.458 m Tris, 0.0104 m citrato. Sistema di tampone JRP = Tampone gel: pH 7.0, 0.009 m Tris, 0.003 m citrato; tampone elettrodo: 0.135 m Tris, 0.040 m citrato. Tutti questi tamponi e i metodi di colorazione utilizzati sono simili a quelli descritti da Steiner e Johnson (1973) e Ayala et al. (1972; 1974a).
Gli alleli in un determinato locus sono stati numerati in base alle mobilità relative dei loro allozimi sui sistemi di tampone elettroforetici specificati. L'allele che produceva l'allozima più frequente è stato designato l.00 e gli alleli corrispondenti a bande che migrano più velocemente o più lentamente sono stati arbitrariamente assegnati valori indicativi delle rispettive mobilità.
Il tampone utilizzato e la notazione per ciascun enzima sono i seguenti: Buffer System A : Esterasi (EST, 1 locus) ; Fosfatasi Alcalina (AP, 1 locus) ; Fosfatasi Acida (ACPH, 1 locus) ; Glutammato Ossaloacetato Transaminasi (GOT, 2 loci) ; Aldeide Ossidasi (AO, 1 locus) ; Deidrogenasi dell'Octanolo (ODH, I locus) ; Deidrogenasi dell'Alcol (ADH, 1 locus) ; Deidrogenasi della Xantina (XDH, 1 locus) ; Aminopeptidasi della Leucina (LAP, 2 loci) ; Deidrogenasi del Glicerolo-3-Fosfato (G-3-PDH, 1 locus). Buffer System C : Enzima Malico (ME, 1 locus) ; Deidrogenasi del Malato (MDH, 2 loci) ; Deidrogenasi dell'α-Glicerofosfato (α-GPDH, 1 locus) ; Fosfoglucomutasi (PGM, I locus) ; Fumarasi (FUM, 1 locus) ; Deidrogenasi del Sorbitolo (SDH, 1 locus) ; Eccokinasi (HK, 2 loci) ; Deidrogenasi dell'Isocitrato (IDH, 1 locus). Buffer System JRP : Deidrogenasi del 6-Fosfogluconato (6-PGDH, 1 locus) ; Deidrogenasi dell'Idrossibutirrato (HBDH, 1 locus) ; Isomerasi del Fosfoglucosio (PGI, 1 locus).
Risultati
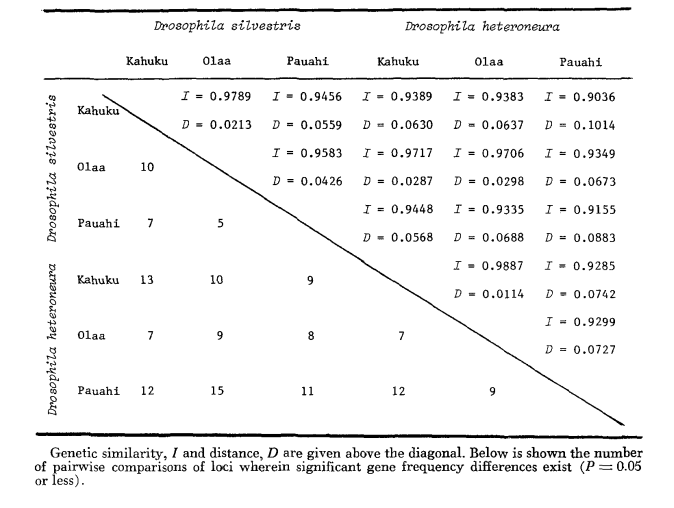
I dati di base sugli allozimi sono forniti nella Tabella 1. La Tabella 2 mostra i risultati dell'uso dei dati nella Tabella 1 per calcolare i coefficienti di somiglianza genetica, I, e distanza, D (Nei 1972) in modo coppia per coppia all'interno e tra le specie. Nella stessa tabella (sotto la diagonale) sono mostrati il numero di loci che mostrano differenze nella frequenza genica (P ≤ 0.05) nei confronti coppia per coppia. Il calcolo è stato effettuato, utilizzando χ2, usando la frequenza dell'allele più comune rispetto a una frequenza aggregata degli altri alleli. È stata utilizzata la correzione di Yates.
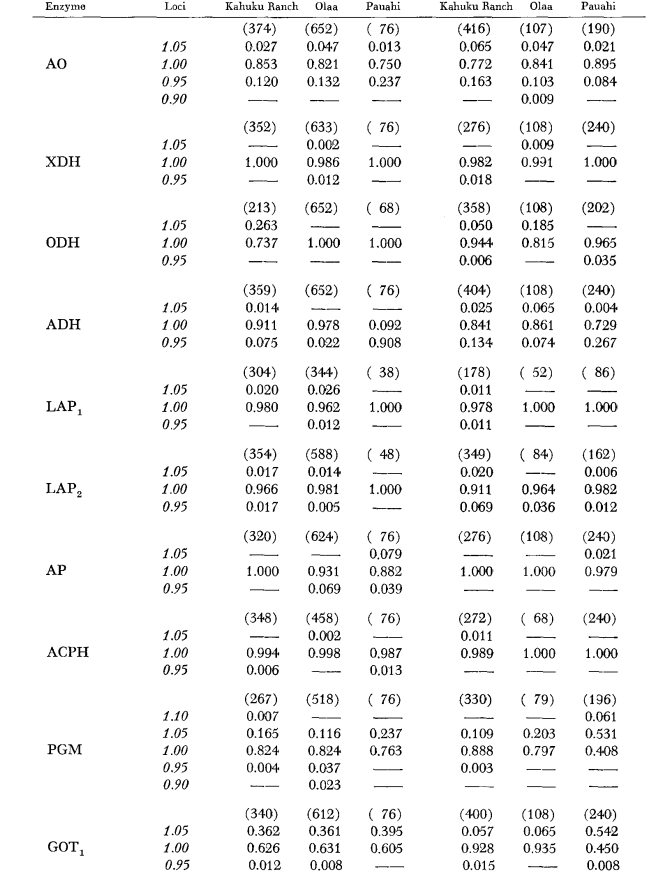
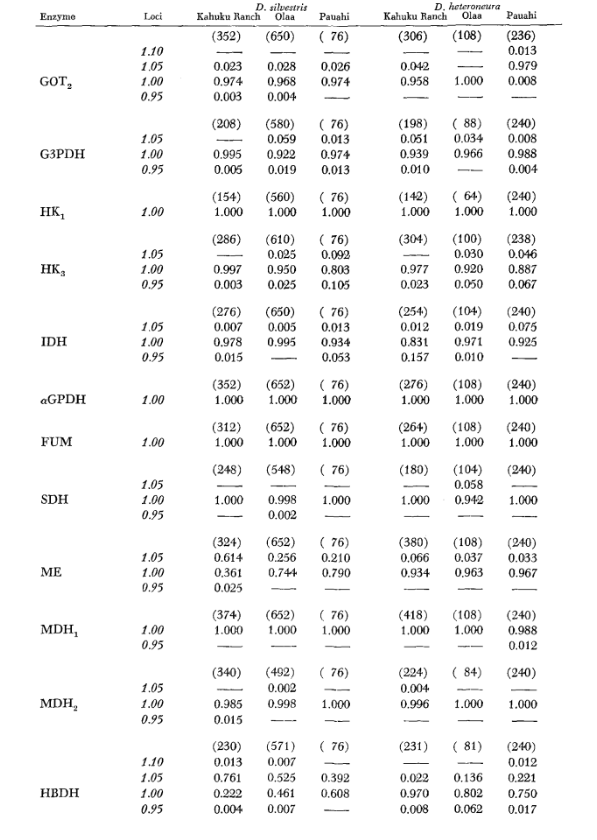
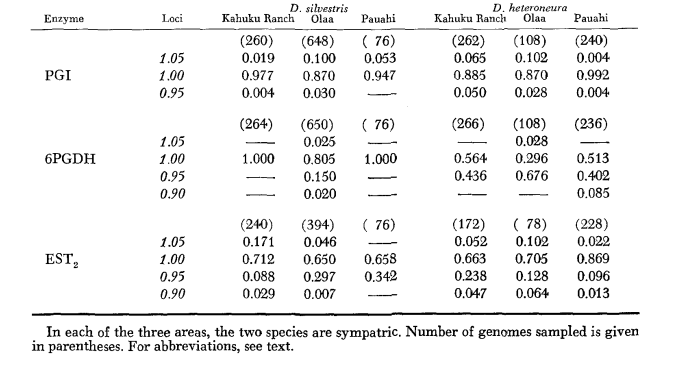
I livelli medi di eterozigosi e le stime della frequenza dei loci polimorfici sono riportati nella Tabella 3. Una rappresentazione grafica della frequenza dell'allele 1.0 per ogni allozima in ciascuna popolazione è presentata nella Figura 2.
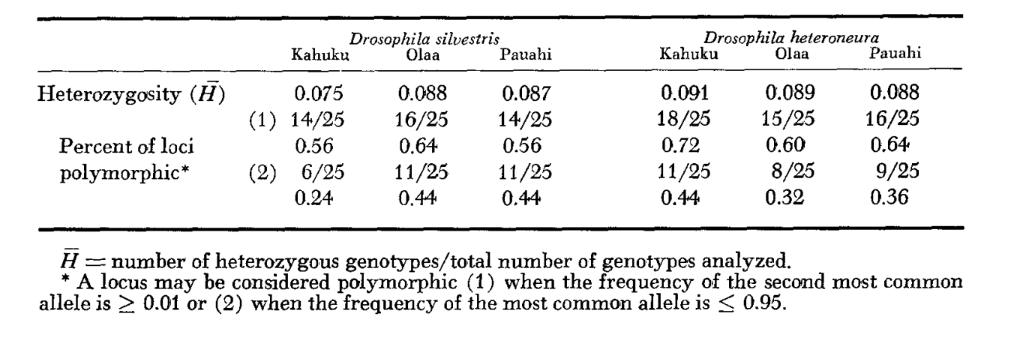
La somiglianza allozimica è molto elevata sia all'interno che tra le specie, sia misurata tramite coefficienti di somiglianza che tramite differenze nella frequenza genica (vedi riepilogo nella Tabella 4). Non ci sono differenze alleliche fisse né all'interno né tra le specie. Nelle tre aree di simpatria, le specie mostrano differenze nella frequenza genica (P = 0.05) in 9, 11 e 13 loci rispettivamente. Questo non è molto diverso dalla variazione all'interno di ciascuna delle specie nelle tre località (silvestris: 5, 7, 10; heteroneura: 7, 9, 12). Le due specie hanno livelli di eterozigosi simili (silvestris 0.083; heteroneura 0.089) e percentuale di loci polimorfici (entrambi 0.37).

Alcuni dettagli sono di interesse. La somiglianza tra le specie è minima a Pauahi (Tabella 2; Figura 2). L'ispezione mostra che ciò è principalmente dovuto alle differenze di frequenza a ADH e GOT2. Grandi differenze a questi loci appaiono tra le specie solo in quest'area; cioè, l'allele 1.00 GOT2 è basso solo in Pauahi heteroneura e l'allele 1.00 di ADH è basso solo in Pauahi silvestris (Tabella 1; Figura 2).
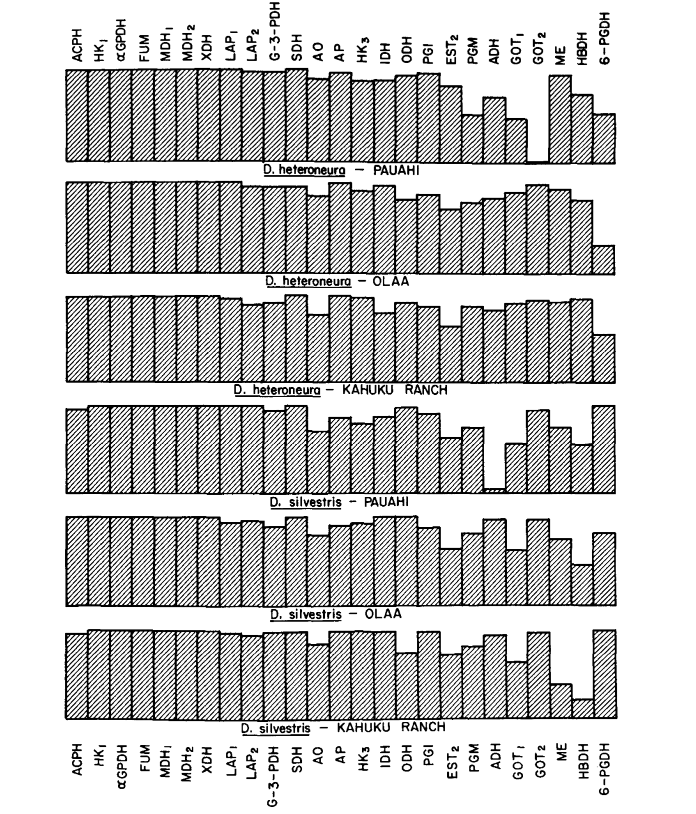
La somiglianza tra le specie è leggermente maggiore nell'area di Kahuku. Qui, le differenze riguardano principalmente GOT1, ME e HBDH. A Olaa la somiglianza tra le specie è massima. GOT1 e HBDH mostrano nuovamente differenze, sebbene non siano così grandi come quelle tra le specie a Kahuku.
Discussione
Le comparazioni genetiche delle popolazioni utilizzando allozimi hanno inondato la letteratura negli ultimi dieci anni (vedi le recensioni di Ayala 1975 e Powell 1975). Poiché sia le proteine studiate che i metodi elettroforetici utilizzati sono stati, nel complesso, simili, i risultati di taxa piuttosto disparati possono essere confrontati utilmente. Ad esempio, la somiglianza genetica tra due specie di lucertole, quando ridotta a un coefficiente come quello di Nei, può essere confrontata direttamente con dati simili provenienti da coppie di specie di topi, piante o Drosophila.
Nel loro articolo seminale, Lewontin e Hubby (1966) hanno evidenziato una serie di motivi per cui i metodi elettroforetici standard saranno inclinati verso una sottostima della variabilità genetica e della differenza genetica. Questo deve essere tenuto presente ancor di più oggi a causa dello sviluppo recente dell'analisi elettroforetica sequenziale. Così Singh, Lewontin e Felton (1976) e Coyne (1976) hanno dimostrato che, per il locus della xantina deidrogenasi di D. pseudo-obscura e persimilis, il numero di alleli in campioni naturali piuttosto piccoli di queste specie è almeno quattro volte maggiore rispetto al numero rivelato dai metodi standard. Per quanto riguarda i confronti interpopolazionali, questi nuovi metodi sembrano certi di ridurre le somiglianze osservate a tutti i livelli di differenziazione della popolazione (ad es., popolazioni locali, sottospecie, semispecie, specie).
Nonostante questi nuovi sviluppi, i confronti basati sui metodi standard continuano a essere preziosi se ne vengono riconosciuti i limiti. D. silvestris e heteroneura sono molto più vicini allozimicamente rispetto a qualsiasi altra coppia di specie di Drosophila finora esaminata, comprese tutte le coppie di specie "sorelle", sottospecie e semispecie (Ayala et al. 1974b). Infatti, i coefficienti di somiglianza per le popolazioni all'interno di silvestris o heteroneura sono dello stesso ordine di grandezza di quelli tra le stesse due specie. Inoltre, le piccole differenze che esistono tra coppie di popolazioni delle due specie non sono le stesse nelle tre aree studiate; cioè, non coinvolgono gli stessi loci o alleli.
Come discusso nell'introduzione, continuiamo a riconoscere queste due entità come buone specie biologiche. Sono morfologicamente distinte, ampiamente simpatiche, comportamentalmente isolate e diverse nei loro polimorfismi cromosomici. Le differenze morfologiche dipendono da circa quattordici poligeni (Val 1976).
Una caratteristica sorprendente e importante di queste specie, tuttavia, è il fatto che gli ibridi reciproci, ottenuti in laboratorio, sono completamente fertili in entrambi i sessi (Craddock 1974; Ahearn e Val 1975). Di conseguenza, deve essere considerata l'ibridazione naturale. Sebbene Craddock (1974) non abbia trovato prove di ciò, Kaneshiro e Val (1977) hanno riportato che circa il due percento delle mosche selvatiche catturate al Kahuku Ranch possono essere identificate come ibridi per mezzo di metodi morfologici. Questa è la stessa regione in cui sono stati trovati ibridi naturali tra due altre specie (Carson, Nair e Sene 1975). L'analisi morfometrica della forma della testa è stata utilizzata con successo per specificare la parentela reciproca esatta di certi individui ibridi F. In un caso, una femmina di D. silvestris catturata in natura ha prodotto una prole composta interamente da ibridi F. Poiché ogni femmina selvatica è stata isolata immediatamente da tutti i maschi al momento della cattura, è chiaro che gli accoppiamenti rilevanti si sono verificati in natura. È importante sottolineare che il background di ampi studi morfometrici sugli ibridi di laboratorio (Val 1976) ha preparato il terreno per queste nuove osservazioni naturali e, di conseguenza, la diagnosi di questi casi si basa su prove comparative solide. Tutti gli individui ibridi naturali sono stati esclusi dai dati elettroforetici presentati in questo articolo.
Un ulteriore fatto che aggiunge alla situazione insolita riguardante queste specie è la loro stretta somiglianza ecologica. Entrambe sono state allevate dallo stesso individuo di steli in decomposizione della comune pianta ospite, Clermontia. Entrambe, inoltre, formano i loro lek in luoghi molto simili, in modo che maschi e femmine di entrambe le specie interagiscano in natura.
Qual è la ragione della grande somiglianza allozimica di queste specie? Si suggeriscono due possibilità. In primo luogo, quando è avvenuta la divergenza tra le specie, potrebbe essere stata accompagnata da una certa quantità di divergenza elettroforetica. Le somiglianze attualmente osservate richiederebbero quindi che tali differenze siano state erose da ibridazione introgressiva successiva alla speciazione. Una seconda opinione sostiene che solo lievi cambiamenti elettroforetici abbiano accompagnato gli eventi di speciazione in primo luogo. Questo potrebbe essere correlato alla notevole vicinanza ecologica.
Sosteniamo la seconda opinione per una serie di motivi. Le due specie sono più simili a Olaa. Tuttavia, forti evidenze di recente ibridazione sono state ottenute solo per Kahuku, dove le specie sono più distanti allozimicamente. A Kahuku, inoltre, le popolazioni naturali non mostrano evidenze di formare un “ibrido swarm” che assomigli alla vasta ricombinazione genetica osservata negli incroci di laboratorio. Infatti, l'introgressione tra le specie sta probabilmente avvenendo, ma potrebbe essere ristretta a uno stretto spettro genotipico a causa della selezione naturale. Così solo geni di importanza biologica minore (il “sistema aperto,” Carson 1975) potrebbero fluire attraverso la barriera delle specie. Al contrario, altri geni potrebbero contrassegnare o essere una parte funzionale di un sistema chiuso di equilibri epistatici unico per una o l'altra delle specie. La selezione potrebbe quindi servire a inibire la rottura di tale “sistema chiuso” (Carson 1975).
A causa della novità geologica dell'isola di Hawaii, siamo tentati di suggerire che D, silvestris e D. heteroneura possano davvero essere recentemente formati nel tempo. Questa circostanza potrebbe essere la causa principale della somiglianza elettroforetica delle due specie. Infatti, potrebbero essere molto più recenti rispetto ai classici “coppie di specie sorelle” (vedi Carson 1976b). Come in altre apparenti neospecie, (ad es. Gottlieb 1974; Turner 1974; Avise, Smith e Ayala 1975), la differenziazione allozimica è stata relativamente lieve. Infatti, esiste la possibilità che la differenza allozimica possa semplicemente correlarsi con il tempo trascorso dall'evento cladistico che ha separato le due entità confrontate (Avise, Smith e Ayala 1975; Carson 1976a). Le differenze morfologiche, d'altra parte, possono servire come migliori indicatori degli effetti sottostanti dei geni regolatori che caratterizzano le importanti differenze genetiche tra le specie recentemente formate.
Autori: F. M. Sene, H. L. Carson
Letteratura citata:
- Ahearn, J. N., H. L. Carson, Th. Dobzhansky e K. Y. Kaneshiro, 1974 Isolamento etologico tra tre specie del sottogruppo planitibia di Drosophila hawaiana. Proc. Natl. Acad. Sci. U.S. 71: 901—903.
- Ahearn, J. N. e F. C. Val, 1975 Ibridi interspecifici fertili di due Drosophila hawaiane simpatiche. Genetics 80: s9.
- Avise, J. C., J. J. Smith e F. J. Ayala, 1975 Differenziazione adattativa con poco cambiamento genico tra due minnows nativi della California. Evolution '29: 411—426.
- AYALA, F. J., 1975 Differenziazione genetica durante il processo di speciazione. Evolutionary Biology 8: 1—78.
- Ayala, F. J., J. R. Powell, M. L. Tracey, C. A. Mourão e S. Pérez-Salas, 1972 Variabilità enzimatica nel gruppo Drosophila willistoni. IY Variazione genica nelle popolazioni naturali di Drosophila willistoni. Genetics 70: 113—139.
- Ayala, F. J., M. L. Tracey, L. G. Barr e J. G. Ehrenfeld, l9f4a Differenziazione genetica e riproduttiva della sottospecie, Drosophila equinoxialis caribbensis. Evolution 28: 24—41.
- Ayala F. J., M. L. Tracey, D. Hedgecock e R. C. Richmond, 1974b Differenziazione genetica durante il processo di speciazione in Drosophila. Evolution 28: 576—592.
- Carson, H. L. 1975 La genetica della speciazione a livello diploide. Am. Naturalist 109: 83—92, 1976a Inferenza del tempo di origine di alcune specie di Drosophila. Nature 259: 395—396, 1976b Differenze genetiche tra specie appena formate. Bioscience 26: 700—701.
- Carson, H. L., P. S. Nair e F. M. Sene, 1975 Ibridi di Drosophila in natura: Prova di scambio genico tra specie simpatiche. Science 189: 806—807.
- Carson, H. L. e H. D. Stalker, 1968 Relazioni tra cromosomi politenici nelle specie hawaiane di Drosophila. II. Il sottogruppo D. planitibia. Univ. Texas Publ. 6818: 355-365.
- Coyne, J. A., 1976 Mancanza di somiglianza genica tra due specie sorelle di Drosophila come rivelato da tecniche varie. Genetics 84: 593—607.
- Craddock, E. M., 1974 Relazioni riproduttive tra specie omosequenziali di Drosophila hawaiana. Evolution 28: 593—606.
- Craddock, E. M. e H. L. Carson, 1975 Variabilità cromosomica in una specie endemica di Drosophila hawaiana. Genetics 80: s23.
- Gottlieb, L. D., 1974 Conferma genetica dell'origine di Clarkia lingulata. Evolution 2B: 244-250.
- Hardy, D. E., 1965 insetti delle Hawaii, Vol. 12. p. 814. University of Hawaii Press, Honolulu.
- Johnson, W. E. e H. L. Carson, 19f5 Variabilità allozimica in Drosophila silvestris. Genetics 80: s46.
- Johnson, W. E., H. L. Carson, K. Y. Kaneshiro, W. W. M. Steiner e M. M. Cooper, 1975 Variazione genetica in Drosophila hawaiana. II. Differenziazione allozimica nel sottogruppo D. planitibia. pp. 563—584. In: Isozymes IV. Genetics and Evolution. A cura di C. L. Markert. Academic Press, New York.
- Kaneshiro, K. Y., 1976 Isolamento etologico e filogenesi nel sottogruppo planitibia di Drosophila hawaiana. Evolution 30: 740-745.
- Kaneshiro, K. Y. e F. C. Val, l977 Ibridazione naturale tra una coppia simpatrica di Drosophila hawaiana. Am. Naturalist (in press).
- Lewontin, R. C. e J. L. Hubby, 1966 Un approccio molecolare allo studio dell'eterozigosi genica nelle popolazioni naturali. II. Quantità di variazione e grado di eterozigosi nelle popolazioni naturali di Drosophila pseudoobscura. Genetics 54: 595—609.
- Nei, M., 1972 Distanza genetica tra popolazioni. Am. Naturalist 106: 283—292.
- Powell, I. R., 1975 Variazione proteica nelle popolazioni naturali di animali. Evol. Biol. 8: 79-113.
- Simon, R. S., R. C. LEWONTin e A. A. Fzczou, 19 Z6 Eterogeneità genetica all'interno degli "alleli" elettroforetici della xantina deidrogenasi in Drosophila pseudoobscura. Genetics 84: 609—629.
- Steiner, W. W. M. e W. E. Johnson, 1973 Tecniche per l'elettroforesi di Drosophila hawaiana. US/IBP Island Ecosystems IRP. Tech. Report 30: ii + 21 pp.
- Turner B. J., 1974 Divergenza genetica delle specie di pesci pupfish della Death Valley: Evidenza biochimica contro evidenza morfologica. Evolution fl8: 281-294.
- Val, F. C., 1976 Genetica delle differenze morfologiche tra due specie interfertili di Drosophila. Genetics 83: s78.