Sulla variabilità del DNA satellitare pBuM189 tra le popolazioni sudamericane di Drosophila buzzatii
Traduzione automatica
L'articolo originale è scritto in lingua EN (link per leggerlo) .
Il DNA satellite pBuM189 è stato analizzato in popolazioni di Drosophila buzzatii che coprono la maggior parte della distribuzione della specie in Sud America. Questo satDNA consiste in monomeri ricchi di A+T di 189 bp e studi precedenti hanno mostrato un rapido tasso di cambiamento evolutivo di questo componente del genoma di D. buzzatii. Un totale di 63 unità di ripetizione pBuM189 provenienti da 14 popolazioni di D. buzzatii (9 dal Brasile e 5 dall'Argentina) sono state studiate. La variabilità nucleotidica media tra le 63 ripetizioni è del 4,2%. Almeno una ripetizione (Juan/4) sembra far parte di un'altra sottocategoria di satDNA pBuM189. L'allineamento nucleotidico di tutte le 63 ripetizioni non ha rivelato sostituzioni nucleotidiche specifiche, né indel, che potessero discriminare ciascuna popolazione o gruppi di popolazioni geograficamente vicine. Tale mancanza di differenziazione interpopolazionale del satDNA è congruente con i dati mtDNA precedenti che indicano un alto flusso genico e pochissima differenziazione delle popolazioni in gran parte della distribuzione di D. buzzatii in Sud America. Il flusso genico potrebbe essere stato possibile durante eventi di glaciazione nel Pleistocene, come quello avvenuto tra 13.000 e 18.000 anni fa, quando D. buzzatii probabilmente aveva una distribuzione più continua rispetto a quella osservata oggi.
La mosca cactofila Drosophila buzzatii (buzzatii cluster, repleta group) ha una vasta distribuzione in Sud America, essendo trovata in gran parte dei territori di Argentina, Bolivia, Paraguay e Brasile. La specie utilizza i tessuti in decomposizione di diversi cactus fico d'India (Opuntia ssp.) e cactus colonnari per nutrirsi e riprodursi (PEREIRA et al. 1983). Negli ultimi duecento anni, i cactus Opuntia sono stati trasportati dall'uomo in diverse regioni del mondo, inclusi i paesi del bacino del Mediterraneo, Africa e Australia. Di conseguenza, D. buzzatii è migrata con i suoi ospiti. Questa situazione ha reso D. buzzatii un organismo appropriato per affrontare domande relative ai cambiamenti genetici durante il processo di colonizzazione (FONTDEVILA et al. 1982; HALLIBURTON e BARKER 1993; ROSSI et al. 1996; FRYDENBERG et al. 2002).
Secondo i dati sulle inversioni cromosomiche, il Chaco argentino è il centro di radiazione più probabile (WASSERMAN 1962; CARSON e WASSERMAN 1965). D. buzzatii è anche molto abbondante nel Chaco, associato a un numero di diverse specie di cactus (Vilela et al. 1980). In generale, le popolazioni brasiliane sono distribuite in modo irregolare in piccole aree di vegetazione xerofitica, dove sono state trovate a frequenze molto basse (VILELA et al. 1983; TIDON-SKLORZ et al. 1994; TIDON-SKLORZ e SENE 1995). L'unica eccezione è il sud del Brasile, dove sono state trovate popolazioni relativamente grandi (VILELA et al. 1983; RUIZ et al. 2000).
È stato postulato che D. buzzatii ha colonizzato il Brasile attraverso cambiamenti nella distribuzione della vegetazione arida durante eventi di glaciazione, come quelli avvenuti alla fine del Pleistocene (BARKER et al. 1985; FIGUEIREDO e SENE 1992).
BAIMAI et al. (1983) hanno analizzato i cromosomi metafasici di diverse popolazioni di D. buzzatii provenienti dall'Argentina e dal Brasile. Tutte le popolazioni campionate mostrano la stessa piastra metafasica. BARKER et al. (1985) non hanno trovato differenze allozimiche significative tra le popolazioni sudamericane. Solo una piccola differenziazione locale sembra essere avvenuta nelle popolazioni brasiliane di Bahia (nord-est) e Tramanda´1 (sud). In un altro studio, FIGUEIREDO e SENE (1992) hanno riportato che delle 16 inversioni cromosomiche rilevate in Argentina, solo due erano presenti nelle popolazioni brasiliane e nessuna è stata trovata nel Brasile nord-orientale. ROSSI et al. (1996) e DE BRITO et al. (2002) hanno trovato alti livelli di uniformità del mtDNA tra le popolazioni sudamericane di D. buzzatii e diversi test hanno indicato un alto flusso genico in gran parte della distribuzione geografica di D. buzzatii . DE BRITO et al. (2002) hanno anche calcolato che D. buzzatii è presente in Brasile da almeno 100 mila anni, suggerendo un'espansione pre-olocenica delle popolazioni argentine di D. buzzatii verso il Brasile.
Per ottenere ulteriori informazioni sulla variabilità genetica di D. buzzatii in Sud America, abbiamo cercato un DNA nucleare. Il DNA satellitare (satDNA) è una classe di DNA non codificante altamente ripetitivo che è disposto in tandem in grandi array omogenei nel genoma di quasi tutti gli organismi eucarioti. Il solitamente rapido tasso di cambiamento evolutivo delle sequenze di satDNA è supportato da un numero di famiglie di satDNA che si sono dimostrate specifiche per le specie (BACH-MANN et al. 1989; KING e CUMMINGS 1997).
Studi precedenti hanno dimostrato che le sequenze di satDNA potrebbero essere utili nella discriminazione delle popolazioni conspecifiche (ELDER e TURNER 1994; WU et al. 1999).
KUHN et al. (1999) hanno descritto il DNA satellitare pBuM189 di D. buzzatii. Consiste di unità di ripetizione leggermente ricche di AT e lunghe 189 bp. Le cinque sequenze pBuM189 descritte sono state ottenute dal DNA genomico di una sola popolazione di D. buzzatii. Esperimenti di ibridazione e PCR non sono riusciti a rilevare sequenze pBuM189 nel genoma di specie strettamente correlate come D. serido, D. borborema o D. koepferae. Pertanto, i dati disponibili indicano un rapido tasso di cambiamento evolutivo di questo componente del DNA nucleare di D. buzzatii.
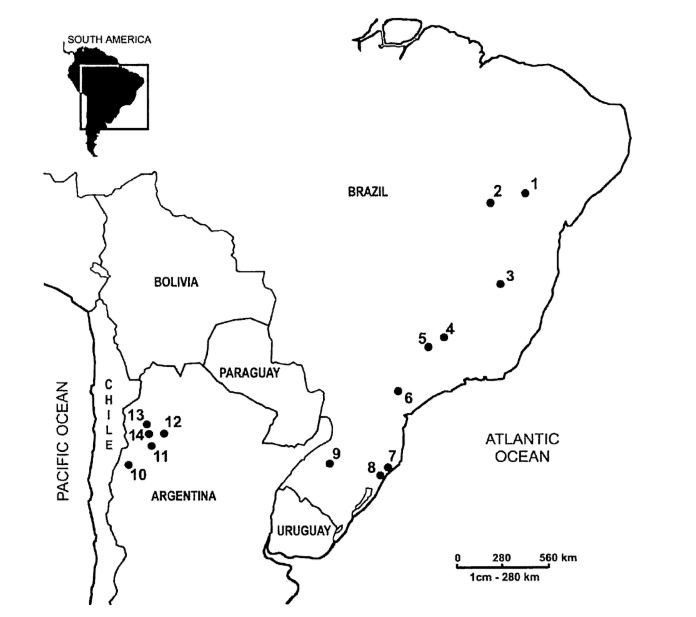
Nello studio attuale, abbiamo analizzato 63 sequenze di pBuM189 da ceppi che rappresentano 14 popolazioni di D. buzzatii (9 brasiliane e 5 argentine). Le popolazioni coprono la maggior parte della distribuzione geografica di questa specie in Sud America.
Materiale e metodi
Campioni
La posizione geografica di tutte le popolazioni di D. buzzatii campionate è mostrata nella Fig. 1. I ceppi utilizzati con le rispettive località sono: D69R5 (1); J79H41 (2); D54F5 (3); H86G8 (4); D42F2 (5); H99X6 (6); H42F1 (7); J25A20 (8); J28E15 (9); ArgE3 (10); Cat (11), Hondo (12); Salta (13) e Ticucho (14).
Tecniche molecolari
Le sequenze pBuM189 sono state isolate originariamente dal genoma di D. buzzatii dopo la digestione del DNA con l'enzima MspI (KUHN et al. 1999). Nel presente lavoro, è stato utilizzato l'enzima SspI (che è presente anche solo una volta per unità di ripetizione pBuM189) per facilitare la procedura di clonazione. I frammenti di satDNA pBuM189 sono stati identificati per la loro apparizione come bande prominenti di circa 190 bp nello sfondo del DNA genomico. Il DNA dalla banda di DNA di 190 bp è stato eluato dal gel mediante incubazione notturna in 500mM NaAc; 1mM EDTA. I frammenti recuperati sono stati ligati in un vettore plasmidico pUC18 e trasformati in cellule competenti di Escherichia coli DH5-a. I cloni ricombinanti sono stati selezionati come colonie bianche su piastre di ampicillina contenenti X-gal (5-bromo-4-cloro-3-indolil-β-D-galattoside) e IPTG (isopropil-β-D-tiogalattopiranosio). La reazione di DNA template per il sequenziamento è stata preparata secondo il manuale del BigDye Terminator Cycle Sequencing Ready Reaction Kit (Perkin-Elmer). Il sequenziamento automatico del DNA è stato eseguito su un sequenziatore ABI Prism™ 377 (Perkin-Elmer).
Il programma MEGA 2.1 (KUMAR et al. 2001) è stato utilizzato per la stima della variabilità nucleotidica, delle distanze genetiche e per la costruzione di un dendrogramma NJ.
Risultati e discussione
Come già previsto dai dati di sequenza precedenti di pBuM189, la digestione del DNA genomico di 13 D. buzzatii ceppi con SspI ha prodotto una banda di DNA fortemente colorata di circa 190 bp, corrispondente ai monomeri di pBuM189, seguita da bande di segnali di intensità decrescente di 380 bp (dimeri), 570 bp (trimeri) e così via. La banda di DNA di 190 bp da ciascuna digestione è stata eluita indipendentemente dal gel e clonata. Un totale di 58 cloni sono stati sequenziati, 39 da 8 popolazioni brasiliane (località 1 – 4, 6 – 9) e 19 da 5 popolazioni argentine (località 10 – 14). L'allineamento nucleotidico di questi 58 cloni con i 5 ripetizioni di pBuM189 (sert/1 – 5 nel presente lavoro) dalla località (5) descritta in precedenza da KUHN et al. (1999) ha dimostrato che rappresentano tutti monomeri di satDNA pBuM189 (Fig. 2).

Le 63 ripetizioni pBuM189 sono leggermente ricche di A+T (71 %) e la variabilità media della sequenza è del 4,2 %. La principale fonte di variazione tra le 63 ripetizioni pBuM189 sono le sostituzioni di nucleotidi singoli, essendo il 69 % specifiche per la ripetizione e le rimanenti comuni a due o più ripetizioni. Sono stati trovati solo 11 indel.
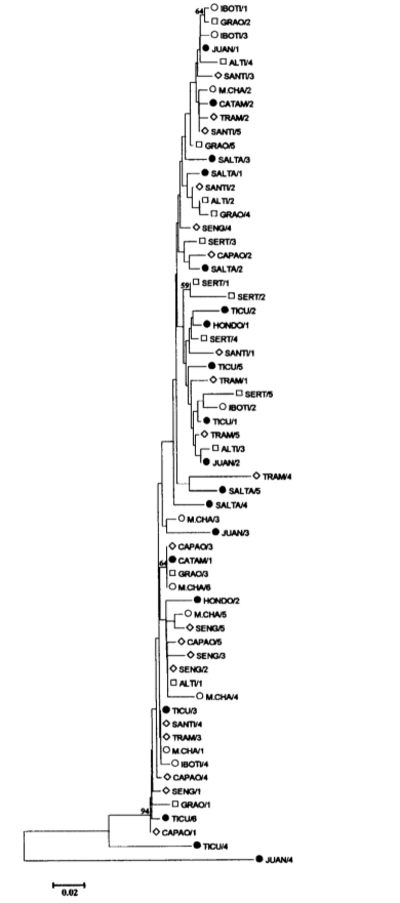
Una ripetizione pBuM189 sembra essere eccezionale. Il clone Juan/4 della località (10) ha mostrato livelli inaspettatamente elevati di divergenza della sequenza in tutti i confronti a coppie (22,1 % in media e sempre superiore al 19 %). Inoltre, mentre le sequenze tipiche di pBuM189 presentano in media l'1,7 % di sostituzioni di nucleotidi specifiche per la ripetizione, ne sono state trovate 30 nella ripetizione Juan/4 (ad es. T nelle posizioni 11 e 42). Un tale alto grado di divergenza nucleotidica suggerisce che la ripetizione Juan/4 potrebbe appartenere a un'altra sottocategoria di pBuM189. Da un punto di vista teorico, l'origine delle sottocategorie di satDNA dipende da variabili come il numero di copie, la distribuzione genomica della famiglia di satDNA e i tassi relativi di scambi non reciproci all'interno e tra cromosomi non omologhi (DOVER 1986).
Se il ripetuto Juan/4 è escluso dal campione, la variabilità nucleotidica media delle rimanenti 62 sequenze pBuM189 diminuisce al 3,7 %. Questo valore è quasi identico al 3,8 % calcolato da KUHN et al. (1999) basato su cinque sequenze pBuM189 isolate da un'unica popolazione di D. buzzatii (località 5).
Tra gli altri ripetuti pBuM189, Ticu/4 dalla località (14) è il più simile a Juan/4 per la presenza di due nucleotidi specifici (G in posizione 70 e A in posizione 88) e un indel (posizione 49) che sono altrimenti esclusivi per il ripetuto Juan/4 e diverse altre sostituzioni in comune con Juan/4 e altri ripetuti (ad es. T in posizione 36 o A in posizione 133). Il ripetuto Ticu/4 stesso è il ripetuto pBuM189 più divergente (10,2 % in media) dopo Juan/4, presentando 6 sostituzioni nucleotidiche specifiche per il ripetuto (posizioni 1, 69, 79, 97, 98, 167 e 188). Pertanto, il Ticu/4 deve essere considerato come un passo evolutivo intermedio tra i ripetuti pBuM189 "tipici" e il ripetuto altamente divergente Juan/4. È interessante notare che entrambi i ripetuti rari Juan/4 e Ticu/4 provengono dall'Argentina, dove la specie probabilmente ha avuto origine (WASSERMAN 1962; CARSON e WASSERMAN 1965).
Secondo dati teorici ed empirici, la differenziazione delle sequenze di satDNA dovrebbe sorgere secondo il concetto di evoluzione concertata (DOVER 1986; ELDER e TURNER 1994). In altre parole, gli array di satDNA provenienti da diverse popolazioni o specie potrebbero essere omogeneizzati per diverse mutazioni a causa dell'effetto di meccanismi molecolari come il crossing-over disuguale e la conversione genica associati all'isolamento genetico e alla deriva genetica.
Allineamento nucleotidico dei 63 ripetizioni di pBuM189 provenienti da 14 popolazioni di D. buzzatii (in media il 4,5 % di ripetizioni per popolazione) non ha rivelato alcuna sostituzione nucleotidica specifica, o indel, che potesse discriminare una popolazione specifica o gruppi di popolazioni geograficamente vicine (Fig. 2). In alcuni casi, più di due cloni condividevano sequenze nucleotidiche identiche, come nel caso dei cloni Capao/3, Grao/3, M.Cha/6 e Catam/1 o M.Cha/1 e Santi/4, ma provengono da località geografiche piuttosto distanti. D'altra parte, non sono state trovate ripetizioni identiche in una singola popolazione.
Le distanze genetiche tra le 63 sequenze nucleotidiche pBuM189 sono state calcolate secondo il metodo dei due parametri di Kimura (KIMURA 1980) e è stato costruito un dendrogramma di "neighbor joining" (SAITOU e NEI 1987) (Fig. 3). La sequenza Juan/4 è stata collocata in un ramo chiaramente separato dagli altri ripetizioni pBuM189, supportando l'assunzione che appartenga a un'altra sottocategoria di satDNA pBuM189. La ripetizione Ticu/4 è stata collocata in un ramo tra la Juan/4 e le rimanenti 61 ripetizioni pBuM189. Tra le rimanenti 61 ripetizioni pBuM189, non è stato osservato alcun cluster contenente sequenze pBuM189 provenienti da una popolazione o gruppi di popolazioni geograficamente vicine.
Nel complesso, i dati indicano alti livelli di somiglianza nella sequenza pBuM189 tra le popolazioni di D. buzzatii che si trovano, in alcuni casi, a più di 3000 km di distanza. Tale mancanza di differenziazione interpopolazionale del satDNA è congruente con i dati mtDNA che indicano un alto flusso genico e molto poca substruttura della popolazione in gran parte della distribuzione di D. buzzatii in Sud America (ROSSI et al. 1996; DE BRITO et al. 2002). Di conseguenza, deve esserci una spiegazione adattativa per la perdita del polimorfismo di inversione cromosomica nelle popolazioni brasiliane (rispetto alle popolazioni argentine), riportata da FIGUEIREDO e SENE (1992).
Attualmente, il Chaco (nord Argentina e est Bolivia) e la Caatinga (nordest Brasile) sono le uniche due ampie aree di vegetazione arida in Sud America, presentando un'alta densità e diversità di specie di cactus. In altre aree, le cactacee sono meno frequenti e distribuite in modo frammentato. Lo stesso vale per le popolazioni di D. buzzatii, che dipendono dai cactus per nutrirsi e riprodursi. È ampiamente accettato che il clima globale abbia subito cicli ricorrenti di tempo freddo-secco e caldo-umido durante il Pleistocene, quando si sono verificati almeno quattro eventi glaciali principali e molti minori. Ad esempio, nell'ultimo evento di glaciazione (da 18.000 a 13.000 anni fa), il clima in Sud America era secco e il Chaco era connesso alle Caatinghe attraverso formazioni xerofitiche nel Brasile centrale e lungo la costa atlantica (AB'SABER 1977). Di conseguenza, D. buzzatii probabilmente aveva una distribuzione molto più continua di quella che si osserva oggi. Tale situazione potrebbe aver consentito un ampio flusso genico tra le popolazioni di D. buzzatii sudamericane.
Ad oggi, la maggior parte dei DNA satellitari descritti nella letteratura è stata isolata da individui di un'unica ceppo o località. I dati presenti sono uno dei pochi esempi che forniscono informazioni sulla variabilità della sequenza di satDNA tra popolazioni naturali di una specie diffusa.
Autori: Gustavo C. S. Kuhn, Fernando F. Franco, Wilson A. Silva Jr, Nilce M. Martinez-Rossi e Fabio M. Sene
Riferimenti:
- Ab'Saber, N. A. 1977. Spazi occupati dall'espansione dei climi secchi dell'America del Sud, in occasione dei periodi glaciali quaternari. – Paleoclimas 3: 1 – 19.
- Bachmann, L., Raab, M. e Sperlich, D. 1989. DNA satellitare e speciazione: un DNA satellitare specifico per specie di Drosophila guanche. – Z. Zool. Syst. Evol. Forsch. 27: 84 – 93.
- Baimai, V., Sene, F. M. e Pereira, M. A. Q. R. 1983. Eterocromatina e differenziazione cariotipica di alcune specie di cactus neotropicali del gruppo Drosophila repleta. – Genetica 67: 81 – 92.
- Barker, J. S. F., Sene, F. M., East, P. P. et al. 1985. Polimorfismo di allozimi e cromosomi di Drosophila buzzatii in Brasile e Argentina. – Genetica 67: 161 – 170.
- Carson, H. L. e Wasserman, M. 1965. Un polimorfismo cromosomico diffuso in una specie diffusa, Drosophila buzzatii. – Am. Nat. 99: 111 – 115.
- De Brito, R. A., Manfrin, M. H. e Sene, F. M. 2002. Filo-geografia del DNA mitocondriale delle popolazioni brasiliane di Drosophila buzzatii. – Genet. Mol. Biol. 2: 161 – 171.
- Dover, G. A. 1986. Spinta molecolare in famiglie multigeniche: come nascono, si diffondono e vengono assimilate le novità biologiche. – Trends Genet. 2: 159 – 165.
- Elder, F. J. e Turner, B. J. 1994. Evoluzione concertata a livello di popolazione: sequenze di DNA satellitare HindIII dei pesci pupfish. – Proc. Natl Acad. Sci. 91: 994 – 998.
- Figueiredo, V. L. C. e Sene, F. M. 1992. Variabilità cromosomica nelle popolazioni brasiliane di Drosophila buzzatii (Diptera, Drosophilidae). – Revta. Bras. Biol. 52: 555 – 561.
- Fontdevila, A., Ruiz, A., Ocana, J. et al. 1982. La storia evolutiva di Drosophila buzzatii. II. Quanto è cambiato il polimorfismo cromosomico nella colonizzazione? – Evolution 36: 843 – 851.
- Frydenberg, J., Pertoldi, C., Dahlgaard, J. et al. 2002. Variazione genetica nelle popolazioni originali e colonizzatrici di Drosophila buzzatii analizzate tramite loci di microsatelliti isolati con un nuovo metodo di screening PCR. – Mol. Ecol. 11: 181 – 190.
- Halliburton, R. e Barker, J. S. F. 1993. Mancanza di variazione del DNA mitocondriale in Drosophila buzzatii australiano. – Mol. Biol. Evol. 10: 484 – 487.
- Kimura, M. 1980. Un unico metodo per stimare il tasso evolutivo delle sostituzioni di basi attraverso studi comparativi delle sequenze nucleotidiche. – J. Mol. Evol. 16: 111 – 120.
- King, L. M. e Cummings, M. P. 1997. La variazione della sequenza di ripetizione del DNA satellitare è bassa in tre specie di scarabei seppellitori del genere Nicrophorus (Coleoptera: Silphidae). – Mol. Biol. Evol. 14: 1088 – 1095.
- Kuhn, G. C. S., Bollgönn, S., Sperlich, D. et al. 1999. Caratterizzazione di un DNA satellitare specifico per specie di Drosophila buzzatii. – J. Zool. Syst. Evol. Res. 37: 109 – 112.
- Kumar, S., Tamura, K., Jakobsen, I. B. et al. 2001. MEGA2: software di analisi genetica evolutiva molecolare. – Arizona State Univ.
- Pereira, M. A. Q. R., Vilela, C. R. e Sene, F. M. 1983. Note sui siti di riproduzione e alimentazione di alcune specie del gruppo repleta del genere Drosophila (Diptera, Drosophilidae). – Ciência e Cultura 35: 1313 – 1319.
- Rossi, M.S., Barrio, E., Latorre, A. et al. 1996. La storia evolutiva di Drosophila buzzatii. XXX. Polimorfismo del DNA mitocondriale nelle popolazioni originali e colonizzatrici. – Mol. Biol. Evol. 13: 314 – 323.
- Ruiz, A., Cansian, A. M., Kuhn, G. C. S. et al. 2000. Il puzzle della speciazione di Drosophila serido: mettere insieme nuovi pezzi. – Genetica 108: 217 – 227.
- Saitou, N. e Nei, M. 1987. Il metodo del neighbor-joining: un nuovo metodo per ricostruire alberi filogenetici. – Mol. Biol. Evol. 4: 406 – 425.
- Tidon-Sklorz, R. e Sene, F. M. 1995. Fauna di Drosophila (Diptera, Drosophilidae) nell'area settentrionale della ‘‘Cadeia do Espinhac¸o'', stati di Minas Gerais e Bahia, Brasile: aspetti biogeografici ed ecologici. – Iheringia Ser. Zool. 78: 85 – 94.
- Tidon-Sklorz, R., Vilela, C. R., Sene, F. M. et al. 1994. Il genere Drosophila (Diptera, Drosophilidae) nella Serra do Cipó, stato di Minas Gerais, Brasile. – Revta. Bras. Ent. 38: 627 – 637.
- Vilela, C. R., Sene, F. M. e Pereira, M. A. Q. R. 1980. Sulla fauna di Drosophila del Chaco e dei pendii orientali delle Ande in Argentina. – Revta. Bras. Biol. 40: 837 – 841. Vilela, C. R., Pereira, M. A. Q. R. e Sene, F. M. 1983. Dati preliminari sulla distribuzione geografica delle specie di Drosophila all'interno dei domini morfoclimatici del Brasile. II. Il gruppo repleta. – Ciên. Cult. São Paulo 35: 66 – 70.
- Wasserman, M. 1962. Studi citologici del gruppo repleta del genere Drosophila. V. Il sottogruppo mulleri. – Univ. Texas Publ. 6205: 85 – 118.
- Wu, W. L., Wang, J. P., Tseng, M. C. et al. 1999. Clonazione e variabilità genetica di un DNA ripetitivo HindIII in Acrossocheilus paradoxus (Cyprinidae). – Genome 42: 780 – 788.